전사인자(transcription factor, TF)는 특정 서열을 지닌 DNA에 결합하여 유전자의 발현을 조절한다. 전사 인자가 결합하는 부위를 transcription factor binding site (TFBS)라고 한다. TFBS는 CHromosome ImmunoPrecipitation (ChIP) 실험을 통해 찾을 수 있다. ChIP는 특정 전사인자를 특이적으로 인지하여 결합하는 항체를 이용하여 전사인자를 농축시키는 실험 방법이다. 이 때 전사인자에 결합되어 있는 genomic DNA도 같이 농축된다. 이렇게 농축된 DNA의 서열을 next generation sequencing (NGS) 기법을 이용해 찾을 수 있다. 이렇게 ChIP 방법과 NGS 기법을 같이 사용하는 것을 ChIP-Seq이라 한다. 이렇게 얻은 서열에서 반복되는 서열 혹은 pattern (sequence motif)은 MEME(Multiple Em for Motif Elicitation)와 같은 프로그램을 이용해 찾을 수 있다.
연습 예제
여기서는 간단한 예제를 이용해 DNA 서열에서 pattern을 찾는 과정을 진행한다.
1. 서열 데이터 확보하기
전사인자 결합 DNA의 서열을 알기위해서는 ChIP-Seq과 같은 실험이 필요하다. 여기서는 실험 결과를 모아둔 ENCODE database에 있는 데이터를 이용한다. 예제로 사용하는 데이터는 전사인지 MYC에 대한 실험결과(https://www.encodeproject.org/experiments/ENCSR784BVD/) 중 500개의 서열만 따로 모은 데이터로 아래와 같이 FASTA 포멧으로 서열 정보가 기입되어 있다.
>NS500343:231:H33YLBGX3:1:11101:17526:1038 1:N:0:TGGGAGT
CCTGANCTAGGTAGTTCCAAAGCTGCACTCCTGGAGCTTTTGCCCTCACAGCTGGCGTGGCTTTTTGGTTAATACT
>NS500343:231:H33YLBGX3:1:11101:24028:1038 1:N:0:TGGGAGT
GGAGGNTGCAGTGAGCCGAGATCGCACCACTGCACTCTAGCCTGGGGGACAGAGCGAGACTCCGTCTCAAAGATCG
>NS500343:231:H33YLBGX3:1:11101:9331:1040 1:N:0:TGGGAGT
CCCTTNCTGCTCACCTGGCAGCAGCTGCTTGTTAGACCCTGGAGGAACTCCAAGAGGAGAGCCACAGAGTCTGACA
>NS500343:231:H33YLBGX3:1:11101:21757:1040 1:N:0:TGGGAGA
CTTTGNGCCTGGGGGAGTTGCACAGGTGAGCTGGGGCCTCACCACTTGCAGCAGGTCAGGAGTTTGGGTCCTAGCA
>NS500343:231:H33YLBGX3:1:11101:13759:1042 1:N:0:TGGGAGT
CGAGGNGCGGGCTCCGGCCTGGGCAAGCGGGTACGCGGCGGAGGCCCCGCAGCGGGGCGGGGAGGGAGGCGTGCGG
Example file
아래 예제 파일을 다운받는다.
2. MEME-Chip을 이용한 motif 찾기
다수의 서열이 지닌 pattern 혹은 sequence motif는 MEME(Multiple Em for Motif Elicitation)와 같은 프로그램을 이용하여 찾을 수 있다. MEME는 web을 통해 실행할 수 있다. 여기서는 ChIP에 특화된 MEME-ChIP를 이용한다. 아래의 과정을 통해 DNA motif를 찾는다.
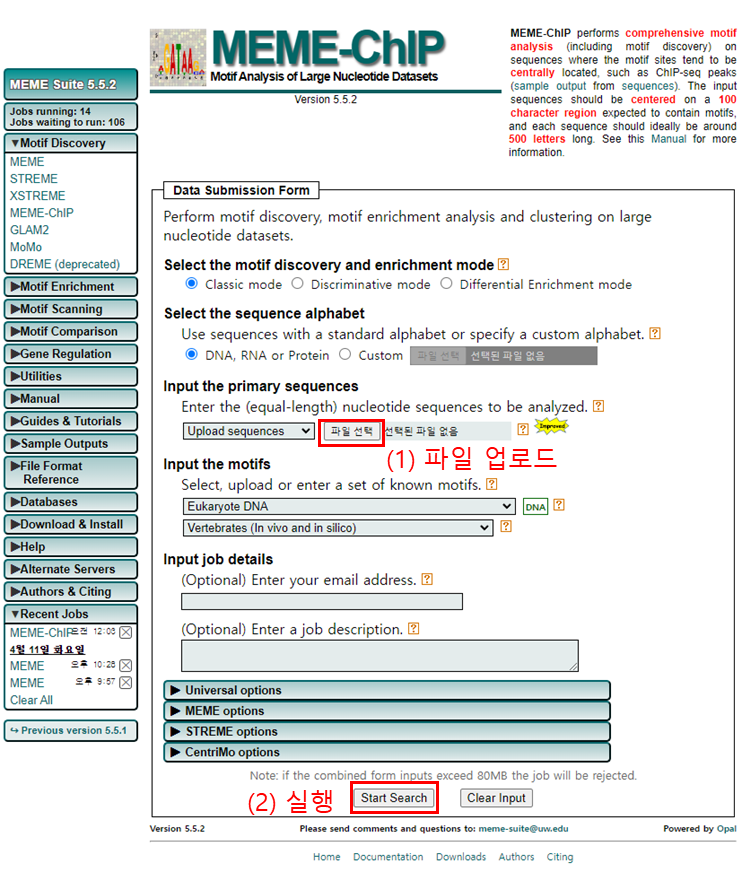
- MEME-ChIP web site에 접속한다
- 파일을 업로드 한다. (MYC_500_reads.txt)
- 실행 버튼(Start Search)를 클릭한다.
- 결과가 나올 때 까지 기다린다.
- 서버 상황에 따라 결과가 나올 때까지 시간이 소요된다.
- URL 주소를 복사하여 보관하면, 시간이 지난뒤 URL을 통해 결과를 확인할 수 있다.
- 다만, 서버에 저장된 결과는 일정시간이 지나면 사라진다. (URL을 통한 접근 불가)
3. Motif 분석하기
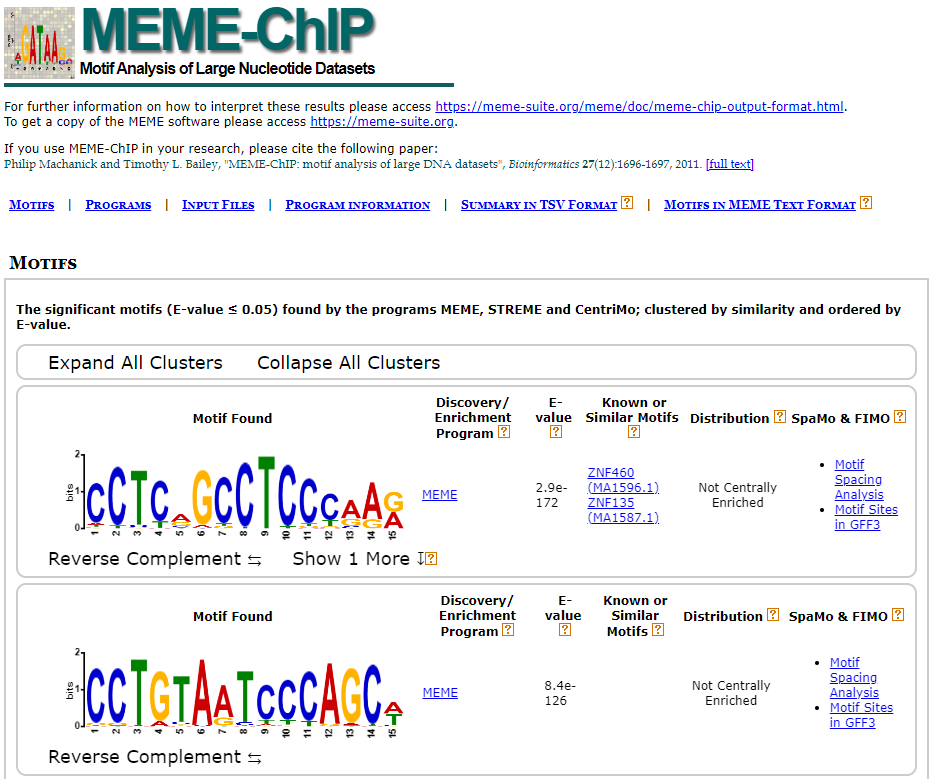
MEME ChIP-Seq 실행 결과 발견한 sequence motif(pattern)를 web server에서 바로 확인할 수 있다. 계산이 완료되면 아래와 같이 화면이 전환된다.

결과 화면에서, MEME-ChIP HTML output link를 클릭하면 아래와 같이 각각의 위치에 따라 특정한 nucleotide가 많이 관찰되는 것을 확인 할 수 있다. DNA 혹은 protein 서열의 pattern을 해당 위치의 글자의 크기로 표현하는 방법을 WebLogo라 하며, 자세한 설명은https://weblogo.berkeley.edu/에서 찾을 수 있다.
총 4개의 motif가 찾아진 것을 확인할 수 있다. 즉, 전사인자 MYC는 아래와 같은 서열에 결합할 가능성이 높다고 할 수 있다.
(*) 하지만, 해당 서열은 ZNF460의 결합 서열이고, cMYC이 결합하는 motif에 해당한다. 파일 준비 과정에 이상이 있었던 것으로 생각한다.