소개
딥러닝과 신약개발 수업 중간 고사 대체 프로젝트입니다.
문헌 조사를 통해 질병이 어떻게 발생하는지 조사하고, 약물 타겟을 찾습니다. 해당 약물 타겟에 결합하는 약물 정보를 ChEMBL에서 찾고 타겟 결합 화합물 데이터셋을 구성합니다. 타겟 결합 예측 deep learning model을 만들고, 타겟 결합 화합물 데이터셋을 이용해 훈련시켜 타겟 결합 약물 예측 모델을 구성합니다. 예측 모델을 화합물 데이터베이스에 적용하여 타겟 결합 화합물을 선정하고, 해당 화합물의 구조와 기능을 표기합니다.
목적
이 과제의 목적은 신약을 위한 초기선도물질(hit) 발굴 과정을 계획하고 실습함으로써 (1) 신약 개발 과정에 대한 이해를 높이고 (2) 실제 신약 개발 과정에서의 전산학적 문제 풀이 능력을 배야하는 것입니다.
과제 수행 방법
중간고사 이전 학습한 내용을 바탕으로, 질병 관련 유전자에 결합할 수 있는 화합물을 예측하는 과제를 수행합니다. 이는 두 개의 JupyterNotebook 작성을 통해 이루어집니다. 1.연구노트.ipynb에는 연구 과정에서 수집한 정보와 데이터 분석 과정을 포함한 code를 기록합니다. 2. 보고서.ipynb에는 연구 결과 전체를 정리하여 다른 사람에게 소개합니다.
과제 수행을 위해 과제 수행 JupyterLab에 접속하고, terminal에서 아래 명령을 실행합니다. 연구를 수행하면서 과정과 결과를 MidTerm_Project 디렉토리의 1. 연구노트.ipynb에 기록합니다. 이 과정에서 사용한 code도 같이 기록합니다. 연구 수행 완료 후 2. 보고서.ipynb에 연구 수행 결과를 정리하는 보고서를 작성합니다.
cd ~
cp ../shared_data/MidTerm_Project . -r연구 진행
연구 수행 및 기록
연구는 1. 질환 선정, 2. 타겟 선정, 3. 데이터 수집, 4. 예측 모델 구성, 5. 신규 화합물 예측의 5 단계로 수행합니다.
이 과정은 1.연구노트.ipynb 에 기록합니다.
1. 질환 선정
1. 질환 선정을 위한 자료를 수집합니다. 문헌을 조사하여 선택한 질환에 관심을 가져야 할 이유를 수집합니다. 질환 유병율이나 질환의 증상의 심각성, 치료 방법의 부재 등의 정보를 수집할 수 있습니다. 수집한 문헌의 출처를 같이 기록해서 해당 정보의 신뢰도를 부여합니다.
- (참고) 문헌 조사시 출처의 신뢰도를 고려합니다. 신뢰도는 논문, 공공기관 (정부기관, 병원) 자료, 뉴스 기사, 웹 문서 순으로 생각할 수 있습니다. 다만, 영문 wikipedia와 같은 웹 문서도 논문을 출처로 명시하고 있다면 신뢰할 만한 자료가 될 수 있습니다. 다만, 이 경우에도 웹 문서 자체보다는 원 출처인 논문을 직접 참고하는 것이 바람직합니다.
- (참고) Ligand-based DTI 예측을 위해서는 target-ligand 상호작용 정보가 필요합니다. 따라서 비교적 많은 연구가 진행되어 있는 질환을 선정하도록 합니다.
2. 질환 치료를 위한 신약 개발 필요성을 설명합니다. 수집한 자료에 근거하여 왜 해당 질환에 대한 신약 개발 필요성을 설명합니다. 해당 내용은 보고서의 소개 부분에서 질환 선정 이유를 설명하기 위해 사용합니다.
2. 타겟 선정
타겟 선정을 위한 자료를 수집합니다. 문헌조사를 통해 질환 치료의 대상이 될 수 있는 유전자 (단백질)을 선정합니다. 해당 질환의 mechanism을 설명하고, 타겟 단백질이 질환 발생과 어떻게 관련 있는지에 집중합니다. 이 정보는 보고서의 소개 부분에서 타겟 선정 이유를 설명하기 위해 사용합니다.
3. 데이터 수집
타겟 결합 화합물 정보를 받고, 데이터 분석을 시작합니다. ChEMBL에서 데이터를 받고, 해당 데이터를 이용해 feature로 전환하는 과정을 수행합니다. (기존 activity 자료를 참고합니다.)
3.1. 타겟 결합 화합물 수집
ChEMBL에서 타겟에 결합하는 화합물 정보를 검색하고 결과를 TSV 형식의 파일로 저장합니다. 저장한 파일은 MidTerm_Project/data 폴더로 복사 (drag & drop)합니다. 이 정보는 positive dataset으로 이용합니다.
학습을 위해서는 Positive dataset 이외에도 negative dataset이 필요합니다. 이는 data/negative_dataset.tsv를 이용하시면 됩니다. 또한 학습된 모델을 이용해 새로운 화합물을 찾을 때는 다양한 화합물에 대한 dataset이 필요한데, 이는 data/chemical_library.tsv 파일을 이용합니다.
(참고) 이 두 데이터는 비교적 분자량이 작고 (200~299), 물과 기름에 비슷한 정도로 녹는 (AlogP = 0.65) 화합물입니다.
- 타겟에 결합하는 화합물 정보 (positive dataset)
- 타겟과 관련 없는 화합물 정보 (negative dataset)
- 화합물 library 정보
3.2. 데이터 로드 (Load data)
수집한 TSV 형식의 데이터를 읽고, 어떤 정보가 있는지 조사합니다. pandas library의 read_csv 함수를 이용합니다.
3.3. Feature 전환
Positive dataset, negative dataset을 읽고 이 데이터의 SMILES 정보를 이용해 molecular fingerprint feature를 계산합니다. 그리고, 이렇게 계산된 feature를 변수 X에 저장하고, positive dataset에는 1, negative dataset에는 0을 부여하여 변수 y를 구성합니다.
4. 예측 모델 구성
예측 모델 구성과정을 설명합니다. Markdown cell에 어떤 예측 모델을 구성할지 설명하고, Code cell을 추가하여 코드를 작성합니다.
Deep neural network 모델을 구성하고, 학습합니다. Activity 2의 code를 참고 합니다. Training (fit) 과정에서 validation_split 을 설정합니다.
- (참고) Cross-validation을 수행하면 좋으나, 편의상 held-out test를 수행합니다. (
validation_split설정)
분석 결과를 그림으로 저장하여 보고서에 활용합니다. 그림은 다음과 같이 savefig 함수를 이용해 저장할 수 있습니다.
fig.savefig('figures/sample.png')5. 타겟 결합 화합물 예측
예측 모델을 화합물 라이브러리 (화합물의 집합)에 적용하여 타겟에 결합하는 화합물을 예측할 수 있습니다.
data/chemical_library.tsv 파일을 pandas.read_csv 함수로 읽고, molecular fingerprint로 전환합니다. (X_library와 같은 변수에 molecular fingerprint를 저장할 수 있습니다.)
딥러닝 모델의 predict 함수를 이용해 label을 예측합니다. (y_library와 같은 값으로 저장합니다.)
마지막으로, 예측 정보를 이용해 가장 점수가 좋은 화합물 10개를 찾고, 그 구조를 비교합니다. 이를 돕기 위해 select_top_10_chemicals 함수를 같이 드렸습니다. 아래와 같이 사용할 수 있습니다.
from src import util
top10 = util.select_top_10_chemicals(chemical_library, y_library)
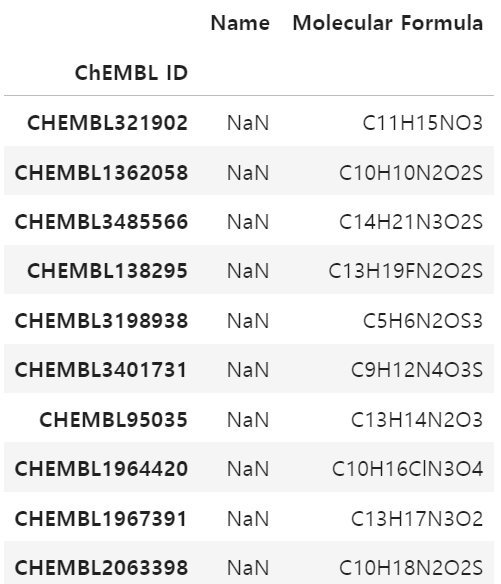
top10아래와 같은 표가 출력됩니다. ChEMBL에서 아래 표의 ChEMBL ID를 이용해 화합물의 구조를 찾아보고, 그 구조 정보를 기록합니다.
평가
연구노트 (50%)
- 과제를 충실히 수행하였는가?
- 수행 과정 기록의 충실도
- 수행 결과 기록의 충실도
보고서 (50%)
- 연구 수행 과정 이해도
- 결과의 체계적 정리 여부
- 내용 전달력
